次世代 DNA 定序的品質管制
隨著次世代定序儀的技術進步,應用領域也日漸廣泛,例如從頭定序,或是分析突變、外顯子體、基因表現等。此外,能夠依據資料分析和資料處理所需的通量,選擇適當模式,更進一步提高這項技術的使用率。不過,要得到良好的定序結果,不論何種應用情境或型號,都需要驗證 NGS 資料庫的大小分佈及濃度。因此使用 NGS 系統時,資訊的品質管制 (QC) 至關重要。
從資料庫製備到 QC,整個程序涵蓋一系列繁瑣的手動操作。本文舉例說明小鼠 RNA 定序分析,使用 MCE-202 MultiNA 自動電泳系統進行 NGS 資料庫 QC,解決操作效率的問題。
MultiNA 應用的定序流程及範圍
在 NGS 資料庫 QC 程序中,MultiNA 的模糊分析軟體* 可用於推算資料庫平均大小、濃度和莫耳濃度。
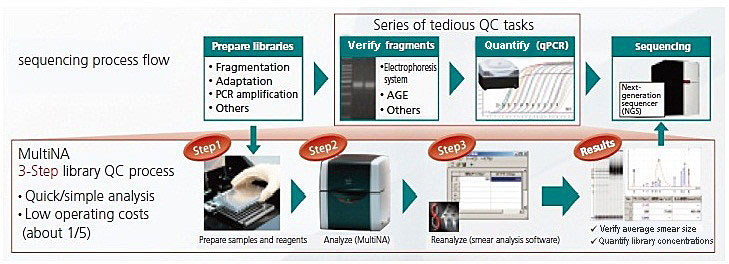
NGS處理流程和MultiNA 在資料庫QC的應用範圍
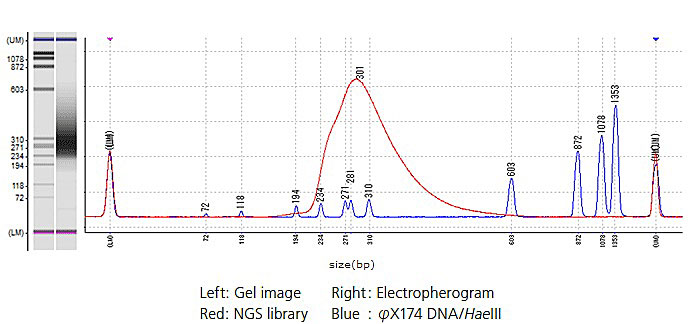
NGS資料庫電泳實例
小鼠 RNA 定序
小鼠 RNA 定序結果顯示,各資料庫之間藉由索引標籤定序,具有足夠的讀取長度及穩定的讀取率。從這些結果可知,使用 MultiNA 進行資料庫 QC,不僅可確認大小分佈,也可在例行分析中同步完成資料庫定量。使用 MultiNA 系統進行資料庫 QC,可快速輕鬆執行 QC。
| Cluster Density | 548 K / mm2 | Total Read Count | 151.5 M reads |
| Read Count (after filtration) |
143.3 M reads | ≥ Q30 ratio | 95.9 % |
| Read Ratio of 4 Libraries | 21.2 - 26.2 % (Four libraries per lane sorted by index tag sequencing: Theoretical value of 25 %) |
||
* MultiNA software requires version 1.12 or later.
Microchip Electrophoresis System for DNA/RNA

使用微晶片的電泳系統,依大小分離 DNA 和 RNA 樣品,因此可以確認核酸 (DNA/RNA) 樣品大小並大致定量。微晶片電泳系統可快速進行電泳分離,螢光檢測器則確保分析的高靈敏度,此外並可全自動完成作業。


