PCR 直接定序的品質檢查
PCR 直接定序技術是直接使用 PCR 反應的擴增產物為模板,以確定鹼基序列,並未進行複製。這是一種非常有效的方法,能夠迅速取得鹼基序列資訊,減少複製後鹼基採用錯誤的影響。
不過,依 PCR 的擴增情況而定,分析結果可能出現很大的差異。要提高成功率,在定序反應之前必須確認 PCR 產物的純度 (品質及含量)。過去確認步驟都是以瓊脂膠體電泳進行,
不過某些情況下,定序分析得到的序列資料,顯示有可能存在密切相關的反應受質,甚至只有單一目標 PCR 產物的時候。之所以出現這種情況,一個可能的原因是瓊脂膠體電泳的靈敏度不足,使用這種方法難以說明主要反應產物中,是否含有微量的 PCR 反應副產物。
此處我們說明 PCR 直接定序的 PCR 產物純度分析,這是日本岡山縣農林水產科學技術中心生物科學研究所,為探索十字花科植物科的管家基因而使用的技術。
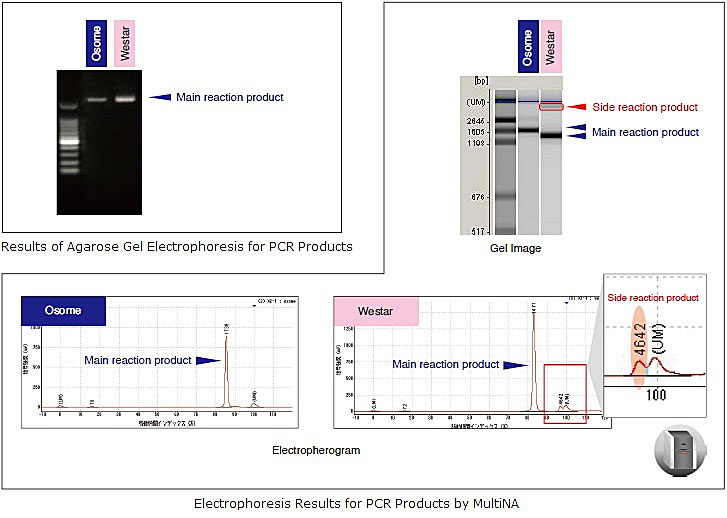
使用瓊脂膠體電泳測得的各種 PCR 產物,大小大致相同,差異並不大。在 MultiNA 的電泳結果中,可在Westar 樣品中測得一個反應副產物,這在膠體影像模式中可以被注意到,而在電泳圖模式中,DNA呈現明顯的峰。不過,瓊脂膠體電泳並未檢測到反應副產物。
使用 MultiNA,可依據電泳圖模式的峰面積計算濃度,此數值足以進行定序反應。因此,MultiNA 對於 PCR 產物純度分析非常有用,是 PCR 直接定序流程的重要工具。
|
* |
本研究使用的樣品和資料,由日本岡山縣農林水產技術中心生物科學研究所 (RIBS) 植物免疫學研究組提供。 |
Microchip Electrophoresis System for DNA/RNA

使用微晶片的電泳系統,依大小分離 DNA 和 RNA 樣品,因此可以確認核酸 (DNA/RNA) 樣品大小並大致定量。微晶片電泳系統可快速進行電泳分離,螢光檢測器則確保分析的高靈敏度,此外並可全自動完成作業。


